随着大型语言模型(LLM)的飞速发展,许多自然语言处理(NLP)任务似乎变得轻而易举。诸如ChatGPT之类的工具有时能生成令人惊叹的优质回复,甚至让经验丰富的专业人士开始思考,某些工作是否会比预期更快地被算法取代。然而,尽管这些模型令人印象深刻,它们在需要精确、领域特定信息提取的任务上仍然面临挑战。
动机:为何构建PICO信息提取器?
构建PICO信息提取器的想法源于一次对话。一位主修国际医疗保健管理的学生,旨在分析帕金森病治疗的未来趋势,并计算如果当前临床试验成功转化为产品,保险公司可能面临的潜在成本。她的第一步是经典的、耗时的工作:从clinicaltrials.gov上发布的试验描述中,手动分离出PICO元素——即“人群”(Population)、“干预措施”(Intervention)、“对照组”(Comparator)和“结局”(Outcome)描述。PICO框架在循证医学中常用于结构化临床试验数据。由于她既不是程序员也不是NLP专家,所有这些工作都完全通过电子表格手动完成。这使得一个事实变得清晰:即使在LLM时代,对生物医学信息提取领域而言,对简单、可靠的工具仍存在实际需求。
步骤1:理解数据并设定目标
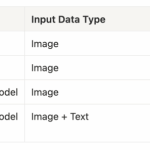
正如所有数据项目一样,首要任务是设定清晰的目标,并明确结果的使用者。在此项目中,目标是提取PICO元素,以便进行后续的预测分析或元研究。目标受众包括所有对系统分析临床试验数据感兴趣的人,无论是研究人员、临床医生还是数据科学家。基于此范围,项目首先从clinicaltrials.gov网站导出JSON格式数据。初步的字段提取和数据清洗提供了部分结构化信息(如表1所示),尤其是针对干预措施,但其他关键字段对于后续的自动化分析来说仍然过于冗长且难以管理。这正是NLP技术发挥作用之处:它能够帮助我们从非结构化文本(如入选标准或测试药物描述)中提炼出关键细节。命名实体识别(NER)技术能够自动化检测和分类关键实体——例如,识别入选标准部分描述的人群组,或在研究摘要中定位结局指标。因此,该项目自然地从基本的预处理过渡到领域适应型NER模型的实施。

表1:从clinicaltrials.gov下载的两个阿尔茨海默病研究的关键信息示例,其中部分信息已从原始数据中提取。(图片作者供稿)
步骤2:评估现有模型
项目的下一步是调研现成的NER模型,特别是那些在生物医学文献上训练并可通过Huggingface(一个Transformer模型中心库)获取的模型。在19个候选模型中,只有BioELECTRA-PICO(1.1亿参数)[1]能够直接用于PICO元素的提取,而其他模型虽然也训练了NER任务,但并非专门针对PICO识别。将BioELECTRA模型应用于一套包含20个手动标注试验的“黄金标准”数据集进行测试,结果显示性能尚可,但远非理想,尤其在“对照组”元素的提取上表现出明显不足。这可能是因为试验摘要中很少直接描述对照组,这促使团队回归到基于实践的规则方法,直接在干预文本中搜索“安慰剂”或“常规护理”等标准对照组关键词。
步骤3:利用领域特定数据进行微调
为进一步提升模型性能,项目转向了微调策略。BIDS-Xu-Lab提供的已标注PICO数据集,包括阿尔茨海默病特定样本[2],为微调提供了可能。为了在追求高准确性的同时,兼顾效率和可扩展性,项目选择了三款模型进行实验。其中,BioBERT-v1.1(1.1亿参数)[3]因其在生物医学NLP任务中的卓越表现,被选作主要模型。此外,还引入了两款更小巧的衍生模型,以优化速度和内存使用:CompactBioBERT是BioBERT-v1.1的精简版本,参数量为6500万;而BioMobileBERT则是一款进一步压缩的变体,仅有2500万参数,并在压缩后进行了额外的持续学习轮次[4]。所有三款模型均利用Google Colab的GPU进行微调,这使得训练过程高效便捷——每款模型在不到两小时内即可完成测试准备。
步骤4:评估与洞察
表2总结了评估结果,揭示了清晰的趋势。所有模型变体在提取“人群”方面表现强劲,其中BioMobileBERT以F1分数0.91领先。对“结局”的提取,所有模型的表现均接近上限。然而,“干预措施”的提取则更具挑战性。尽管召回率较高(0.83–0.87),但精确率相对滞后(0.54–0.61),模型经常在自由文本中标记出额外的药物提及——这通常是因为试验描述会提及背景信息中的药物或“类似干预”的关键词,但并非必然聚焦于计划中的主要干预措施。
更深入的检查突显了生物医学NER的复杂性。“干预措施”有时以短小、零碎的字符串形式出现,如“使用整体”、“周”、“顶部”或“含有组织”,这些对于试图理解研究列表的研究人员来说价值甚微。同样,对“人群”的检查也产生了令人沮丧的例子,如“百分比”或“州”,这表明需要额外的清理和管道优化。与此同时,模型也能够提取出令人印象深刻的详细人群描述,例如“符合条件的、被诊断为认知未受损、或可能患有阿尔茨海默病、额颞叶痴呆或路易体痴呆的成年人”。虽然此类长字符串可能是正确的,但对于实际的摘要来说往往过于冗长,因为每个试验的参与者描述都非常具体,通常需要某种形式的抽象或标准化。
这强调了生物医学NLP中的一个经典挑战:上下文至关重要,领域特定文本往往难以通过纯粹的通用提取方法处理。对于“对照组”元素,基于规则的方法(匹配明确的对照组关键词)表现最佳,这提醒我们,将统计学习与实用启发式方法相结合,往往是实际应用中最可行的策略。
这些“误导性”提取的一个主要来源,源于试验在更广泛的背景部分中的描述方式。未来,可能的改进包括增加一个后处理过滤器以丢弃短小或模糊的片段,整合领域特定的受控词汇(从而只保留已识别的干预术语),或应用概念链接到已知本体。这些步骤有助于确保提取管道生成更清晰、更标准化的输出。
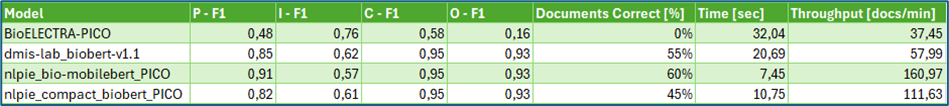
表2:PICO元素提取的F1分数,所有PICO元素部分正确文档的百分比,以及处理时长。(图片作者供稿)
关于性能:对于任何最终用户工具而言,速度与准确性同等重要。BioMobileBERT的紧凑尺寸使其推断速度更快,使其成为首选模型,尤其是在“人群”、“对照组”和“结局”元素的提取上表现最优。
步骤5:实现工具可用性——部署
技术解决方案的价值体现在其可访问性。最终的提取管道被封装在一个Streamlit应用程序中,允许用户上传clinicaltrials.gov数据集,在不同模型间切换,提取PICO元素,并下载结果。快速摘要图表能够一目了然地展示主要的干预措施和结局(参见图1)。为使用户能够比较性能耗时,从而体会到较小架构带来的效率提升,性能相对较低的BioELECTRA模型也被特意保留在工具中。尽管该工具的推出未能让先前进行手动数据提取的学生节省时间,但希望它能惠及其他面临类似任务的研究者。
为简化部署过程,该应用程序已通过Docker进行容器化,以便追随者和合作者能够迅速启动并运行。同时,GitHub仓库 [5]也投入了大量精力进行完善,提供了详尽的文档,以鼓励进一步的贡献或针对新领域的改编。
经验总结
该项目展示了开发一个真实世界信息提取管道的完整历程——从设定清晰的目标、评估现有模型,到利用专业数据进行微调,并最终部署一个用户友好的应用程序。尽管模型和数据均已具备,但将它们转化为真正有用的工具,其挑战远超预期。处理复杂、多词的生物医学实体,这些实体往往仅能被部分识别,凸显了“一刀切”解决方案的局限性。提取文本中缺乏抽象性也成为识别全局趋势的障碍。展望未来,需要更专注的方法和管道优化,而非仅仅依赖现成的解决方案。
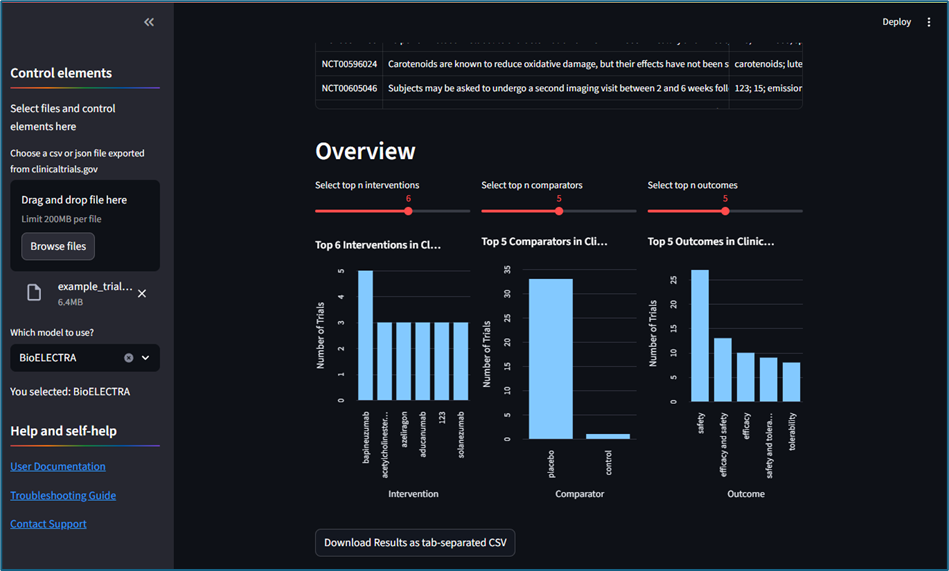
图1:运行BioMobileBERT和BioELECTRA进行PICO提取的Streamlit应用程序的输出示例。(图片作者供稿)
如果对扩展此工作或将此方法应用于其他生物医学任务感兴趣,欢迎探索GitHub仓库 [5]并做出贡献。只需分叉项目,祝您编程愉快!