大型语言模型(LLM)的兴起,让许多自然语言处理(NLP)任务看似轻而易举。ChatGPT等工具有时能生成令人惊叹的优质回复,甚至让经验丰富的专业人士开始思考,某些工作是否会比预期更快地由算法接管。然而,尽管这些模型表现出色,在需要精确的领域特定信息提取任务上,它们仍面临挑战。
动机:为何构建PICO信息提取器?
构建PICO提取器的想法源于一次与一位国际医疗保健管理专业毕业生的交流。这位学生当时正在分析帕金森病治疗的未来趋势,并试图计算如果当前临床试验成功转化为产品,保险公司可能面临的潜在成本。这项工作的第一步是传统且耗时的:从clinicaltrials.gov上发布的临床试验描述中,手动分离PICO要素——即人群(Population)、干预措施(Intervention)、对照组(Comparator)和结果(Outcome)的描述。PICO框架在循证医学中常用于结构化临床试验数据。由于该学生既非编程人员也非NLP专家,她完全依靠手工操作,通过电子表格完成这项工作。这促使人们认识到,即使在大型语言模型时代,市场对生物医学信息提取领域依然存在对简单、可靠工具的真实需求。
第一步:理解数据并设定目标
如同任何数据项目,首要任务是设定清晰的目标并明确结果的使用者。在此项目中,目标是提取PICO要素,以支持后续的预测分析或元研究。目标受众包括所有对系统分析临床试验数据感兴趣的人员,无论是研究者、临床医生还是数据科学家。基于此范围,项目最初从clinicaltrials.gov导出了JSON格式的数据。初步的字段提取和数据清洗提供了一些结构化信息(如表1所示),尤其是在干预措施方面,但其他关键字段对于后续自动化分析而言仍然过于冗长且难以管理。这正是NLP技术大显身手之处:它能够从非结构化文本(如入选标准或测试药物描述)中提炼出关键细节。命名实体识别(NER)技术可以自动检测并分类关键实体——例如,识别资格部分中描述的人群组,或在研究摘要中精确定位结果测量。因此,项目自然从基本预处理阶段过渡到领域自适应NER模型的实施。
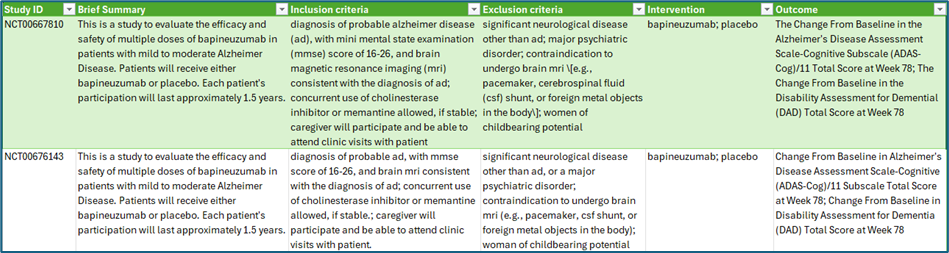
表1:从clinicaltrials.gov网站下载的两项阿尔茨海默病研究的关键信息元素(作者供图)
第二步:基准测试现有模型
接下来,研究团队对现成的命名实体识别(NER)模型进行了调研,特别是那些在生物医学文献上进行训练并通过Hugging Face(Transformer模型的核心存储库)提供的模型。在19个候选模型中,只有BioELECTRA-PICO(1.1亿参数)[1]能够直接用于提取PICO要素,而其他模型虽然经过NER任务训练,但并非专门针对PICO识别。在研究团队自行构建的包含20项手动标注试验的“黄金标准”数据集上测试BioELECTRA,结果显示性能尚可但远非理想,尤其在“对照组(Comparator)”元素的提取上表现出明显不足。这可能是因为对照组在试验摘要中很少被详细描述,因此不得不回归到一种实用的基于规则的方法,即直接在干预文本中搜索“安慰剂”或“常规护理”等标准对照组关键词。
第三步:使用领域特定数据进行微调
为进一步提升性能,项目转向了模型微调,这得益于BIDS-Xu-Lab提供的已标注PICO数据集,其中包含针对阿尔茨海默病症的样本[2]。为了在追求高准确性的同时兼顾效率和可扩展性,研究人员选择了三款模型进行实验。其中,拥有1.1亿参数的BioBERT-v1.1[3]因其在生物医学NLP任务中的卓越表现而被选作主要模型。此外,还引入了两款更小、派生而来的模型,以优化速度和内存占用:6500万参数的CompactBioBERT是BioBERT-v1.1的精简版本;而仅有2500万参数的BioMobileBERT则是进一步压缩的变体,它在压缩后又经过了一轮持续学习[4]。所有这三款模型均利用Google Colab的GPU进行了微调,从而实现了高效训练——每款模型在不到两小时内即可完成测试准备。
第四步:评估与洞察
表2中总结的评估结果揭示了清晰的趋势。所有模型变体在人群(Population)提取方面表现强劲,其中BioMobileBERT以F1分数0.91领先。结果(Outcome)提取在所有模型中都接近最优水平。然而,干预措施(Interventions)的提取则更具挑战性。尽管召回率(recall)较高(0.83–0.87),但精确率(precision)相对滞后(0.54–0.61),模型经常在自由文本中标记出额外的药物提及——这通常是因为试验描述中提及的药物或“类干预”关键词旨在描述背景信息,而非必然聚焦于计划中的主要干预措施。
通过仔细检查,这凸显了生物医学命名实体识别的复杂性。干预措施有时以短小、零碎的字符串形式出现,如“使用整体”、“周”、“顶部”或“组织与”,这些对于试图理解研究列表的研究者来说价值甚微。同样,对人群的检查也产生了一些令人深思的例子,如“百分之几”或“具有……的州”,这表明需要额外的清理和管道优化。与此同时,模型也能提取出令人印象深刻的详细人群描述,例如“符合条件的认知正常成人,或可能患有阿尔茨海默病、额颞叶痴呆或路易体痴呆的成人”。虽然这些长字符串可能是正确的,但对于实际的总结来说往往过于冗长,因为每个试验的参与者描述都非常具体,通常需要某种形式的抽象或标准化处理。
这强调了生物医学NLP中的一个经典挑战:上下文至关重要,领域特定文本常常抵制纯粹的通用提取方法。对于对照组(Comparator)元素,基于规则的方法(匹配明确的对照组关键词)效果最佳,这提醒我们,在实际应用中,将统计学习与实用启发式方法相结合,通常是最可行的策略。
这些“不良”提取的一个主要来源,是由于试验在更广泛的背景描述部分中的表述方式。未来可能的改进措施包括:添加后处理过滤器以丢弃短小或模棱两可的片段;整合领域特定的受控词汇表(从而只保留已识别的干预术语);或应用概念链接到已知本体论。这些步骤有助于确保管道生成更清晰、更标准化的输出。
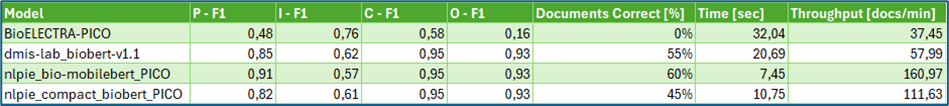
表2:PICO元素提取的F1分数,所有PICO元素部分正确文档的百分比,以及处理时长(作者供图)
关于性能:对于任何最终用户工具而言,速度与准确性同等重要。BioMobileBERT的紧凑尺寸带来了更快的推理速度,使其成为首选模型,尤其是在人群、对照组和结果元素方面表现最佳。
第五步:提升工具可用性——部署
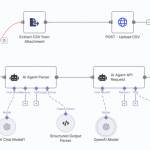
技术解决方案的价值体现在其可访问性上。最终的管道被封装在一个Streamlit应用程序中,允许用户上传clinicaltrials.gov数据集,在不同模型之间切换,提取PICO元素并下载结果。快速摘要图提供了主要干预措施和结果的概览(参见图1)。为使用户能够比较不同模型的性能时长,从而体会到小型架构带来的效率提升,研究团队特意保留了性能相对较弱的BioELECTRA模型。尽管该工具的推出未能让那位学生免去数小时的手动数据提取工作,但希望它能惠及其他面临类似任务的人。
为了使部署过程简单明了,该应用程序已通过Docker进行容器化,以便感兴趣的用户和合作者能够快速启动并运行。GitHub仓库[5]也投入了大量精力进行文档编写,以鼓励进一步的贡献或在新领域进行适配。
经验教训
该项目展示了开发一个真实世界信息提取管道的完整历程——从设定明确目标、基准测试现有模型,到在专业数据上微调模型,并最终部署一个用户友好的应用程序。尽管用于微调的模型和数据都已准备就绪,但将其转化为一个真正有用的工具却比预期更具挑战性。处理复杂、多词的生物医学实体(这些实体往往只能被部分识别)凸显了“一刀切”解决方案的局限性。提取文本中缺乏抽象性也成为了任何旨在识别全球趋势的人的障碍。展望未来,需要更专注的方法和管道优化,而非仅仅依赖简单的“即插即用”解决方案。
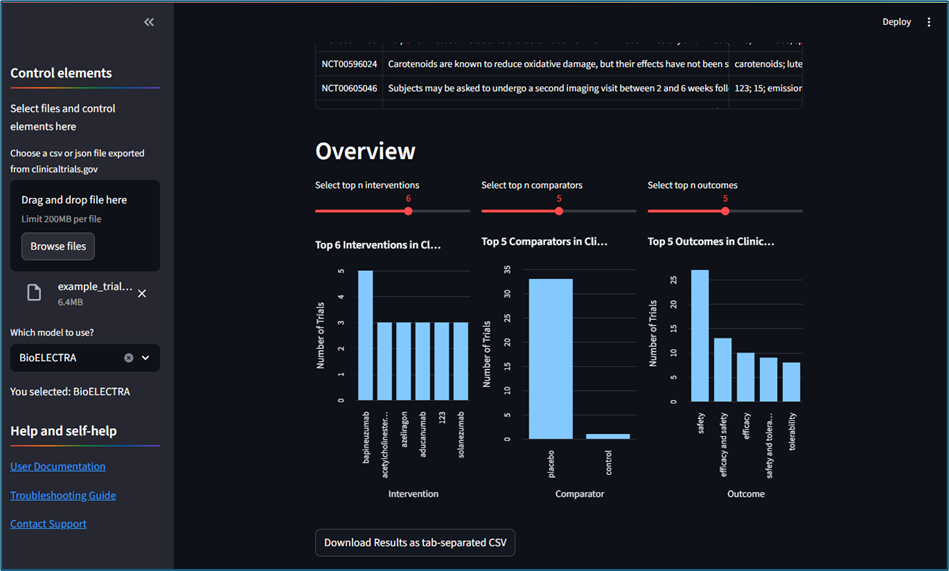
图1:运行BioMobileBERT和BioELECTRA进行PICO提取的Streamlit应用输出示例(作者供图)
如果您对扩展这项工作或将此方法应用于其他生物医学任务感兴趣,欢迎探索GitHub仓库[5]并贡献代码。请您克隆项目并享受编程的乐趣!